Białka kolca wirusa SARS-CoV-2 upośledzają naprawę uszkodzeń DNA i hamują rekombinację V(D)J in vitro
Źródło: Viruses. 2021 Oct; 13(10): 2056. SARS–CoV–2 Spike Impairs DNA Damage Repair and Inhibits V(D)J Recombination In Vitro
Streszczenie
Drugi koronawirus ciężkiego ostrego zespołu oddechowego (ang. severe acute respiratory syndrome coronavirus 2, SARS-CoV-2) doprowadził do wybuchu pandemii koronawirusa (COVID-19) w 2019 roku, która negatywnie wpłynęła na zdrowie publiczne i światową gospodarkę. Odporność nabyta odgrywa kluczową rolę w zwalczaniu infekcji wirusem SARS-CoV-2 i bezpośrednio wpływa na wyniki kliniczne pacjentów. Badania kliniczne wskazują, że pacjenci z ostrym przebiegiem COVID-19 wykazują opóźnioną i słabą odpowiedź immunologiczną nabytą; jednakże mechanizm, za pomocą którego SARS-CoV-2 hamuje odporność pozostaje niejasny. W niniejszym opracowaniu, stosując linię komórkową in vitro, informujemy, że białka kolca tego wirusa znacząco utrudniają naprawę uszkodzeń DNA, która jest niezbędna dla skutecznej rekombinacji V(D)J w odporności nabytej. W sposób mechanistyczny ustaliliśmy, że białka kolca umiejscawiają się w jądrze i blokują naprawę uszkodzeń DNA utrudniając przyjmowanie do uszkodzonego miejsca najważniejszych białek BRCA1 uczestniczących w naprawie DNA. Nasze ustalenia ujawniają potencjalny mechanizm molekularny, za pomocą którego białka kolca mogą hamować odporność nabytą i uwidaczniać ewentualne skutki uboczne szczepionek opartych o białka kolca pełnej długości.
Czy białko kolcowe SARS-Cov-2 powoduje katastrofalne uszkodzenia DNA?
Stabilizowanie kodu – mRNA i receptory Toll-podobne – dr Mike Williams
Korzyści i ryzyka z terapii genowej na C-19 wśród nastolatków – dr Robert Malone
Białko kolcowe SARS-Cov-2 jest bardzo niebezpieczne, jest cytotoksyczne.
Wytyczne Dotyczące Przedklinicznych Badań Farmakologicznych i Toksykologicznych Szczepionek – EMA
1. Wprowadzenie
Drugi koronawirus ciężkiego ostrego zespołu oddechowego (SARS-CoV-2) jest odpowiedzialny za trwającą od 2019 roku pandemię koronawirusa (COVID-19), która pociągnęła za sobą ponad 2,3 miliony zgonów. SARS-CoV-2 to wirus RNA otoczkowy z jedną nicią sensowną, który składa się z białek strukturalnych i niestrukturalnych[1]. Po zakażeniu wspomniane białka wirusowe przejmują kontrolę i rozregulowują mechanizm komórkowy gospodarza, aby replikować, gromadzić i szerzyć wirusy potomne[2]. Przeprowadzone niedawno badania kliniczne wykazały, że infekcja wirusem SARS-CoV-2 ma niebywały wpływ na liczbę i funkcjonowanie białych krwinek [3.4.5.6]. W porównaniu z ozdrowieńcami, u których przebieg choroby był łagodny lub umiarkowany, u pacjentów z ostrą postacią COVID-19 występuje znacząco niższą łączna liczba limfocytów T, limfocytów T pomocniczych i limfocytów T supresorowych[3,4]. Dodatkowo COVID-19 opóźnia powstawanie IgG oraz IgM po wystąpieniu objawów[5,6]. Powyższe obserwacje kliniczne łącznie sugerują, że wirus SARS-CoV-2 oddziałuje na nabytą odpowiedź immunologiczną. Jednak mechanizm, za pomocą którego tłumi odporność nabytą pozostaje niejasny.
Jako dwa kluczowe systemy nadzoru gospodarza, układ odpornościowy i system naprawy DNA są głównymi układami, na których polegają organizmy wyższe, kiedy trzeba bronić się przed rozmaitymi zagrożeniami i dla utrzymania homeostazy tkanek. Pojawiają się dowody, które wskazują, że te dwa systemy są wzajemnie zależne, zwłaszcza podczas rozwoju i dojrzewania limfocytów[7]. Jako jedna z najważniejszych ścieżek naprawy dwuniciowych pęknięć nici DNA (ang. double-strand DNA break, DSB), naprawa poprzez scalanie niehomologicznych końców DNA (ang. non-homologous end joining, NHEJ) pełni decydującą rolę w specyficznej dla limfocytów rekombinacji V(D)J zależnej od endonukleaz genu aktywującego rekombinację (ang. RAG, recombination-activating gene), co skutkuje wysoce zróżnicowanym wachlarzem przeciwciał w receptorach limfocytów B i T (ang. T cell receptors, TCRs) w limfocytach T[8]. Dla przykładu utrata funkcji kluczowych białek naprawczych DNA takich jak ATM, DNA-Pkcs, 53BP1 i innych prowadzi do wad naprawy NHEJ, które powstrzymują produkcję limfocytów funkcjonalnych B i T doprowadzając do niedoboru odpornościowego[7,9,10,11]. W przeciwieństwie do tego infekcja wirusowa wywołuje zazwyczaj uszkodzenie DNA za pomocą różnych mechanizmów takich jak indukcja wytwarzania reaktywnych form tlenu (ang. reactive oxygen species, ROS) i stres replikacyjny w komórkach gospodarza[12,13,14]. Jeśli uszkodzenie DNA nie może zostać prawidłowo naprawione, przyczynia się do rozszerzenia patologii wywołanej zakażeniem wirusowym. Z tej przyczyny za cel postawiliśmy sobie zbadanie czy białka wirusa SARS-CoV-2 przejmują kontrolę nad systemem naprawczym uszkodzeń DNA, tym samym negatywnie wpływając na odporność adaptacyjną in vitro.
2. Materiały i metody
2.1. Przeciwciała i odczynniki
DAPI (nr katalogowy MBD0015), doksorubicyna (numer katalogowy D1515), H₂O₂ (nr katalogowy H1009) oraz przeciwciała przeciwko ß-tubulinie (nr katalogowy T4026) zakupione zostały od firmy Sigma-Aldrich. Przeciwciała przeciwko znacznikowi polihistydynowemu (nr katalogowy 2698), H2A (nr katalogowy 12349), H2A.X (nr katalogowy 7631), ɣ-H2A.X (nr katalogowy 2577), Ku80 (nr katalogowy 2753) i Rad51 (nr katalogowy 8875) zostały zakupione od firmy Cell Signaling Technology (Danvers, Massachusetts, USA). Przeciwciała przeciwko 53BP1 (nr katalogowy NB100-304) i RNF168 (nr katalogowy H00165918-M01) pozyskano od firmy Novus Biologicals (Novus Biologicals, Littleton, Kolorado, USA). Przeciwciała przeciwko laminatowi B (nr kat. 374015), ATM (nr kat. 135663), kinazie białkowej DNA-PK (nr kat. 5282) i białkom BRCA1 (nr kat. 28383) zostały zakupione od firmy Santa Cruz Biotechnology (Santa Cruz, Kalifornia, USA). Przeciwciała przeciwko białkom XRCC4 (nr kat. PA5-82264) zakupiono od firmy Thermo Fisher Scientific (Waltham, Massachusetts, USA).
2.2. Plazmidy
Plazmidy pHPRT-DRGFP i pCBASceI podarowała nam uprzejmie Maria Jasin (plazmidy z repozytorium plazmidów non-profit Addgene nr 26476 i 26477) [15]. Plazmidy pimEJ5GFP były darem od Jeremy’ego Starka (plazmidy Addgene nr 44026) [16]. Białka NSP1, NSP9, NSP13, NSP14, NSP16, białka kolca oraz białka nukleokapsydu zostały najpierw syntetyzowane na drodze optymalizacji kodonów, a następnie sklonowane do wektora ekspresyjnego ssaków pUC57 z C-końcowym znacznikiem 6xHis. Sekwencja sygnałowa rekombinacji (ang. recombination signal sequences, RSS) ze wstawką 12-nukleotydową, odwrócona sekwencja komplementarna białek GFP i RSS ze wstawką 23-nukleotydową zostały syntetyzowany do wektora reporterowego V(D)J. Następnie sekwencja ta została sklonowana do wektora pBabe-IRES-mRFP, aby wygenerować wektor reporterowy pBabe–12RSS–GFPi–23RSS–IRES–mRFP. Sekwencja RSS ze wstawką 12-nukleotydową: 5′–CACAGTGCTACAGACTGGAACAAAAACC–3′. Sekwencja RSS ze wstawką 23-nukleotydową: 5′–CACAGTGGTAGTACTCCACTGTCTGGCTGTACAAAAACC–3′. Konstrukty ekspresyjne RAG1 i RAG2 stanowiły hojny dar Martina Gellerta (plazmidy Addgene 13328 i 13329)[17].
2.3. Komórki i kultura komórkowa
Komórki HEK293T i HEK293 pozyskane z American Type Culture Collection (ATCC) zostały wyhodowane w atmosferze 5% CO₂ w temperaturze 37 ºC na pożywce Dulbecco’s modified Eagle’s medium (DMEM, wysoki poziom glukozy, GlutaMAX) (Life Technologies, Carlsbad, Kalifornia, USA) zawierającej serum płodowe z cieląt w stężeniu objętościowym (v/v) 10% (ang. fetal calf serum, FCS, Gibco), penicylinę w stężeniu objętościowym 1% (v/v) (100 IU/mL) i streptomycynę (100 μg/mL). Komórki reporterowe HEK293T–DR–GFP i HEK293T–EJ5–GFP zostały wygenerowane tak jak w poprzednim opisie i wyhodowane w atmosferze 5% CO₂ w temperaturze 37 ºC na wyżej wspomnianym podłożu hodowlanym.
2.4. Testy reporterowe HR i NHEJ
Naprawa HR i NHEJ w komórkach HEK293T została zmierzona tak jak w poprzednim opisie przy użyciu stabilnych komórek DR-GFP i EJ5-GFP. W skrócie, komórki reporterowe stabilne 0.5 x 10⁶ HEK293T wysiano na płytki 6-dołkowe i transfekowano 2 μg plazmidów ekspresji I-SceI (pCBASceI) wraz z plazmidami ekspresji białek SARS-CoV-2. Czterdzieści osiem godzin po transfekcji i zastosowaniu aspiryny, komórki zostały zebrane i zbadane techniką cytometrii przepływowej pod kątem ekspresji komórek GFP. Średnie zostały uzyskane z trzech odrębnych eksperymentów.
2.5. Frakcjonowanie komórek i metoda immunoblottingu
Do analizy frakcji komórkowych wykorzystano zestaw Subcellular Protein Fractionation Kit (Thermo Fisher) zgodnie z zaleceniami producenta. Lizaty białek zostały określone ilościowo przy użyciu zestawu BCA reagent (Thermo Fisher Scientific, Rockford, Illinois, USA). Białka zbadano techniką elektroforezy w żelu poliakrylamidowym w obecności laurylosiarczanu sodu (ang. sodium dodecyl sulfate–polyacrylamide gel electrophoresis, SDS–PAGE). Białka zostały przeniesione na błony nitrocelulozy (Amersham protran, 0.45 μm NC) i przeanalizowane metodą immunoblottingu przy użyciu specyficznych przeciwciał pierwszorzędowych, a następnie przeciwciał drugorzędowych sprzężonych z HRP. Pasma białek wykryto przy użyciu zestawu SuperSignal West Pico lub Femto Chemiluminescence kit (Thermo Fisher Scientific).
2.6. Test kometowy
Komórki traktowano różnymi reagentami uszkodzeń DNA, a następnie zebrano je we wskazanych punktach czasowych do analizy. Z komórek (1 x 10⁵ komórek/mL w zimnej solance buforowanej fosforanami [PBS]) odtworzono zawiesinę w 1-procentowym roztworze niskotopliwej agarozy w temperaturze 40 ºC w stosunku 1:3 obj./obj. i wlano ją za pomocą pipety na slajdy CometSlide. Slajdy zostały następnie zanurzone w uprzednio schłodzonym buforze do lizy (1.2 M NaCl, 100 mM EDTA, 0.1% laurylosarkozynianu sodu, 0.26 M NaOH pH> 13) na trwającą całą noc (18-20 h) lizę w temperaturze 4 ºC w ciemności. W dalszej kolejności slajdy ostrożnie wyjęto i zatopiono w buforze płuczącym (0.03 M NaOH i 2 mM EDTA, pH > 12) w temperaturze pokojowej (RT) na 20 minut w ciemności. Krok polegający na płukaniu powtórzono dwukrotnie. Slajdy zostały następnie przeniesione do pionowej komory do elektroforezy zawierającej bufor płuczący i rozdzielone na 25 min przy napięciu 0.6 V/cm. Na koniec slajdy umyto w wodzie destylowanej, zabarwiono 10 μg/mL jodku propidyny i zbadano pod mikroskopem fluorescencyjnym. Dwadzieścia pól z około 100 komórkami w każdej próbce zostało ocenionych i określonych ilościowo przy użyciu oprogramowania Fiji w celu określenia długości ogona (ruchów ogona).
2.7. Immunofluorescencja
Komórki zostały wysiane na szkiełku nakrywkowym na płytce 12-dołkowej i transfekowane wskazanymi plazmidami przez 24 h. Następnie były traktowane lub nie były traktowane reagentami uszkodzeń DNA zgodnie z układem doświadczalnym. Komórki utrwalano w 4-procentowym roztworze paraformaldehydu (PFA) w PBS przez 20 min w RT, a następnie permeabilizowano je w 0,5-procentowym roztworze Tritonu X-100 przez 10 min. Szkiełka zablokowano w 5-procentowym roztworze koziej surowicy (NGS) oraz inkubowano przeciwciałami pierwszorzędowymi rozcieńczonymi w 1-procentowym roztworze NGS przez całą noc w temperaturze 4 ºC. Próbki były następnie inkubowane wskazanymi przeciwciałami drugorzędowymi oznaczonymi Alexa Fluor 488 lub 555 (Invitrogen) rozcieńczonymi w 1-procentowym roztwierze NGS w RT przez 1 h. W dalszej kolejności były barwione DAPI przez 15 min w RT. Szkiełka nakrywkowe zostały zamontowane za pomocą Dako Fluorescence Mounting Medium (Agilent) i obejrzane pod mikroskopem konfokalnym firmy Nikon (Eclipse C1 Plus). Całość oceny została przeprowadzona w warunkach ślepej próby.
2.8. Analiza rekombinacji V(D)J
W skrócie, plazmidy reporterowe V(D)J zawierają odwrócone sekwencje białek GFP oraz IRES, które napędzają podlegające stałej ekspresji białka RFP. Podlegające stałej ekspresji RFP służą jako wewnętrzna kontrola transfekcji. Po współtransfekcji genów aktywujących rekombinację ½ (RAG1/2) do komórek, RAG1/2 przecinają RSS i zmodyfikowaną indukcję DSBs, a jeżeli występuje rekombinacja V(D)J, odwrócone GFPs są podwiązywane w kolejności pozytywnej przez naprawę NHEJ. Wówczas komórki wykazują ekspresję funkcjonalnych GFP. A zatem komórki podwójnie dodatnie GFP i RFP stanowią odczyt testu reporterowego V(D)J[18]. Komórki 293T przy 70% konfluencji zostały transfekowane samym białkiem reporterowym GFP rekombinacji V(D)J (tło) lub w połączeniu z konstruktami ekspresyjnymi RAG1 i RAG2 w stosunku 1µg białka reporterowego GFP rekombinacji V(D)J: 0.5 µg RAG1: 0.5 µg RAG2. Następnego dnia zmieniono pożywkę i po upływie dodatkowych 48 h, komórki zostały zebrane i zbadane techniką cytometrii przepływowej pod kątem ekspresji GFP i RFP.
2.9. Analiza statystyczna
Wszystkie doświadczenia powtórzono co najmniej trzykrotnie stosując niezależenie zebrane lub przygotowane próbki. Dane zostały przeanalizowane przy użyciu metody ANOVA lub testu Studenta, a następnie testów wielokrotnego porównania Tukey’a z wykorzystaniem programu GraphPad 8.
3. Wyniki
3.1. Wpływ białek wirusa SARS-CoV-2 zlokalizowanych w jądrze na naprawę uszkodzeń DNA
Naprawa uszkodzeń DNA zachodzi głównie w jądrze dla zagwarantowania stabilności genomu. Mimo że białka wirusa SARS-CoV-2 są syntezowane w cytosolu[1], niektóre białka wirusów można także wykryć w jądrze, w tym Nsp1, Nsp5, Nsp9, Nsp13, Nsp14 i Nsp16[19]. Zbadaliśmy czy białka tego wirusa umiejscowione w jądrze wywierają wpływ na system naprawy uszkodzeń DNA komórki gospodarza. W tym celu utworzyliśmy plazmidy ekspresyjne białek wirusa wraz z plazmidami ekspresyjnymi kolca i nukleoproteidu, które ogólnie uznaje się za białka zlokalizowane w cytosolu. Potwierdziliśmy ich ekspresję i lokalizację stosując techniki immunoblottingu oraz immunofluorescencji (Ilustracja 1A i Ilustracja S1A). Uzyskane wyniki były zgodne z wynikami wcześniejszych badań[19]; białka Nsp1, Nsp5, Nsp9, Nsp13, Nsp14 i Nsp16 są rzeczywiście umiejscowione w jądrze, zaś nukleoproteidy są na ogół zlokalizowane w cytosolu. Nieoczekiwanie odkryliśmy duże zagęszczenie białek kolca w jądrze (Ilustracja 1A). Naprawa NHEJ i proces rekombinacji homologicznej (HR) to dwie główne ścieżki naprawy DNA, które nie tylko stale monitorują i zapewniają integralność genomu, ale są również fundamentalne dla funkcjonowania komórek odporności nabytej[9]. Aby ocenić czy wspomniane białka wirusa zakłócają ścieżkę naprawy DSB, przeanalizowaliśmy proces naprawy miejscowo specyficznego DSB wywołanego przez endonukleazy I-SceI przy użyciu białka bezpośredniej fluorescencji z zielonym powtórzeniem (DR-GFP) oraz całkowitych systemów reporterowych NHEJ-GFP (EJ-5-GFP) odpowienio dla HR i NHEJ[15,16]. Nadekspresja Nsp1, Nsp5, Nsp13, Nsp14 i białek kolca zmniejszyła efektywność zarówno naprawy HR, jak i NHEJ (Ilustracja 1B-E i Ilustracja S2A,B). Oprócz tego ustaliliśmy także, że nadekspresja Nsp1, Nsp5, Nsp13 i Nsp14 radykalnie tłumiła proces rozmnażania w porównaniu z innymi zbadanymi białkami (Ilustracja S3A,B). Z tej przyczyny hamujący wpływ Nsp1, Nsp5, Nsp13 i Nsp14 na naprawę uszkodzeń DNA może być spowodowany drugorzędnymi skutkami takimi jak zatrzymanie wzrostu i śmierć komórki. Co ciekawe, białka kolca ulegające nadekspresji nie oddziaływały na morfologię komórki ani na proces jej rozmnażania, lecz znacząco blokowały naprawę zarówno HR, jak i NHEJ (Ilustracja 1B-E i Ilustracje S2A,B oraz S3A,B).

Wpływ białek drugiego koronawirusa ciężkiego ostrego zespołu oddechowego (SARS-CoV-2) zlokalizowanych w jądrze na naprawę uszkodzeń DNA. (A) Rozmieszczenie subkomórkowe białek wirusa SARS-CoV-2. Immunofluorescencja została przeprowadzona po upływie 24 h po transfekcji plazmidów wyrażających białka wirusa do komórek HEK293T. Podziałka liniowa: 10 µm. (B) Schemat białek reporterowych EJ5-GFP wykorzystywanych do monitorowania procesu scalania niehomologicznych końców DNA (NHEJ). (C) Wpływ pustego wektora (E.V) i białek wirusa SARS-CoV-2 na proces NHEJ służący naprawie uszkodzeń DNA. Wartości oznaczają średnie ± standardowe odchylenie (ang. standard deviation, SD) z trzech niezależnych eksperymentów (patrz reprezentatywne wykresy przedstawiające analizę metodą cytometrii przepływowej na Ilustracji S2A). (D) Schemat białek reporterowych DR-GFP wykorzystywanych do monitorowania procesu rekombinacji homologicznej (HR). (E) Wpływ E.V i białek wirusa SARS-CoV-2 na naprawę uszkodzeń DNA metodą HR. Wartości oznaczają średnie ± SD z trzech niezależnych eksperymentów (patrz reprezentatywne wykresy przedstawiające analizę metodą cytometrii przepływowej na Ilustracji S2B). Wartości oznaczają średnie ± SD, n = 3. Istotność statystyczną określono stosując test jednoczynnikowej analizy wariancji (ang. analysis of variance, ANOVA) dla (C,E). ** p < 0.01, *** p < 0.001, *** p < 0.0001.
3.2. Białka kolca wirusa SARS-CoV-2 hamują naprawę uszkodzeń DNA
Ponieważ białka kolca są kluczowe dla częściowego determinowania wniknięcia wirusa do komórek gospodarza i znajdują się w centrum zainteresowania strategii stosowanych przy opracowywaniu większości szczepionek[20,21], zbadaliśmy bardziej dogłębnie rolę, jaką białka kolca pełnią w naprawie uszkodzeń DNA i związanej z nią rekombinacji V(D)J. Zazwyczaj uważa się, że białka kolca są syntezowane na siateczce śródplazmatycznej szorstkiej (ang. endoplasmic reticulum, ER)[1]. W następstwie modyfikacji potranslacyjnych takich jak glikozylacja, białka kolca przemieszczają się poprzez aparat błony komórkowej wraz z innymi białkami wirusa, aby stworzyć dojrzały wirion[1]. Białko kolca składa się z dwóch głównych podjednostek, S1 i S2, a także z kilku domen funkcjonalnych lub powtórzeń[22]. (Ilustracja 2A). W stanie natywnym białka kolca istnieją jako nieaktywne białka pełnej długości. W czasie infekcji wirusowej, proteazy komórek gospodarza takie jak furyna aktywują białka kolca rozdzielając je na podjednostki S1 i S2, co jest konieczne, by wirus mógł wniknąć do komórki docelowej[23]. Zbadaliśmy dokładniej rozmaite podjednostki białek kolca w celu wyjaśnienia właściwości funkcjonalnych koniecznych, by mogło dojśc do powstrzymania procesu naprawy DNA. Jedynie białka kolca pełnej długości silnie blokowały zarówno ścieżkę naprawy NHEJ, jak i HR (Ilustracja 2B-E i Ilustracja S4A,B). Następnie staraliśmy się ustalić czy białka kolca bezpośrednio przyczyniają się do niestabilności genomowej poprzez hamowanie procesu naprawy DSB. Monitorowaliśmy poziomy DSBs z wykorzystaniem testów kometowych. Po zastosowaniu różnych zabiegów służących naprawie uszkodzeń DNA, takich jak promieniowanie gamma, zastosowanie doksorubicyny i H₂O₂, proces naprawy w obecności białek kolca odbywa się na mniejszą skalę (Ilustracja 2F,G). Dane te łącznie dowodzą, że białka kolca bezpośrednio wpływają na naprawę DNA w jądrze.

Białka kolca drugiego koronawirusa ciężkiego ostrego zespołu oddechowego (SARS-CoV-2) powstrzymują naprawę uszkodzeń DNA. (A) Diagram pierwotnej struktury białka kolca wirusa SARS-CoV-2. Podjednostka S1 obejmuje domenę N-końcową (ang. N-terminal domain, NTD, 14-305 pozostałości) oraz domenę wiążącą receptory (ang. receptor-binding domain, RBD, 319-541 pozostałości). Podjednostka S2 składa się z peptydu fuzji (ang. fusion peptide, FP, 788-806 pozostałości), powtórzonej sekwencji heptapeptydów 1 (ang. heptapeptide repeat sequence, HR1, 912-984 pozostałości), HR2 (1163-1213 pozostałości), domeny transbłonowej (ang. transmembrane domain, TM, 1213-1237 pozostałości) i domeny cytoplazmatycznej (ang. cytoplasm domain, CT, 1237-1273 pozostałości). (B,C) Wpływ stopniowo zwiększanej ekspresji białek kolca na naprawę uszkodzeń DNA w komórkach HEK-293T. (D,E) Jedynie białka kolca pełnej długości hamują naprawę uszkodzeń DNA przez scalanie niehomologicznych końców DNA (NHEJ) i rekombinację homologiczną (HR). Wartości oznaczają średnie ± SD z trzech niezależnych eksperymentów (patrz reprezentatywny wykres przedstawiający badanie metodą cytometrii przepływowej na Ilustracji S4A,B). (F) Komórki HEK293T transfekowane białkami kolca pełnej długości (S-FL) wykazywały więcej uszkodzeń DNA niż komórki transfekowane pustym wektorem, S1, i S2 w innych warunkach uszkodzeń DNA. Dla doksorubicyny: 4 µg/mL, 2 h. Dla promieniowania gamma: 10 Gy, 30 min. Dla H2O2: 100 µM, 1 h. Podziałka liniowa: 50 µm. (G) Analogiczne określenie ilości momentów ogona komety z 20 różnych dziedzin, przy czym n > 200 komet z trzech niezależnych doświadczeń. Istotność statystyczną oceniono stosując test jednoczynnikowej analizy wariancji (ANOVA). Nieznaczna istotność statystyczna (ang. Not Significant, NS): * p < 0.05, ** p < 0.01, *** p < 0.001, **** p < 0.0001.
3.3. Białka kolca zakłócają pozyskiwanie białek punktu kontrolnego naprawiających uszkodzenia DNA
W celu potwierdzenia obecności białek kolca w jądrze, przeprowadziliśmy analizę frakcji subkomórkowej i ustaliliśmy, że białka kolca są wzbogacone nie tylko we frakcjach błony komórkowej, ale występują także w znaczącej liczbie we frakcjach jądrowych, a nawet możliwe jest wykrycie objawów ich obecności we frakcji związanej z chromatyną (Ilustracja 3A). Zaobserwowaliśmy również, że białko kolca ma trzy różne formy – wyższe pasmo to wysoce glikowane białko kolca, środkowa to białko kolca pełnej długości, zaś niższa to rozdzielona podjednostka białka kolca. Zgodnie z wynikami testu kometowego, wykryliśmy ponadto zwiększenie ekspresji markera uszkodzenia DNA, γ–H2A.X, w komórkach z nadekspresją białek kolca w warunkach uszkodzenia DNA (Ilustracja 3B). Niedawno przeprowadzone badanie sugeruje, że białka kolca wywołują stres ER i degradację białek związaną z ER [24]. Aby wykluczyć możliwość, że białka kolca blokują naprawę DNA przyczyniając się do degradacji białek naprawczych uszkodzeń DNA, sprawdziliśmy ekspresję niektórych kluczowych białek naprawczych uszkodzeń DNA w ścieżkach naprawy NHEJ i HR i ustaliliśmy, że wspomniane białka naprawcze były stabilne w następstwie nadekspresji białek kolca (Ilustracja 3C). Celem ustalenia w jaki sposób białka kolca powstrzymują zarówno szlaki naprawy NHEJ, jak i HR, poddaliśmy analizie proces rekrutacji BRCA1 i 53BP1, które są kluczowymi białkami punktu kontrolnego odpowiednio dla naprawy HR i NHEJ. Ustaliliśmy, że białka kolca znacząco blokowały formowanie się ognisk zarówno BRCA1, jak i 53BP1 (Ilustracja 3D-G). Łącznie dane te wskazują, że białka kolca pełnej długości wirusa SARS-CoV-2 hamują naprawę uszkodzeń DNA utrudniając pozyskiwanie białek naprawczych DNA.
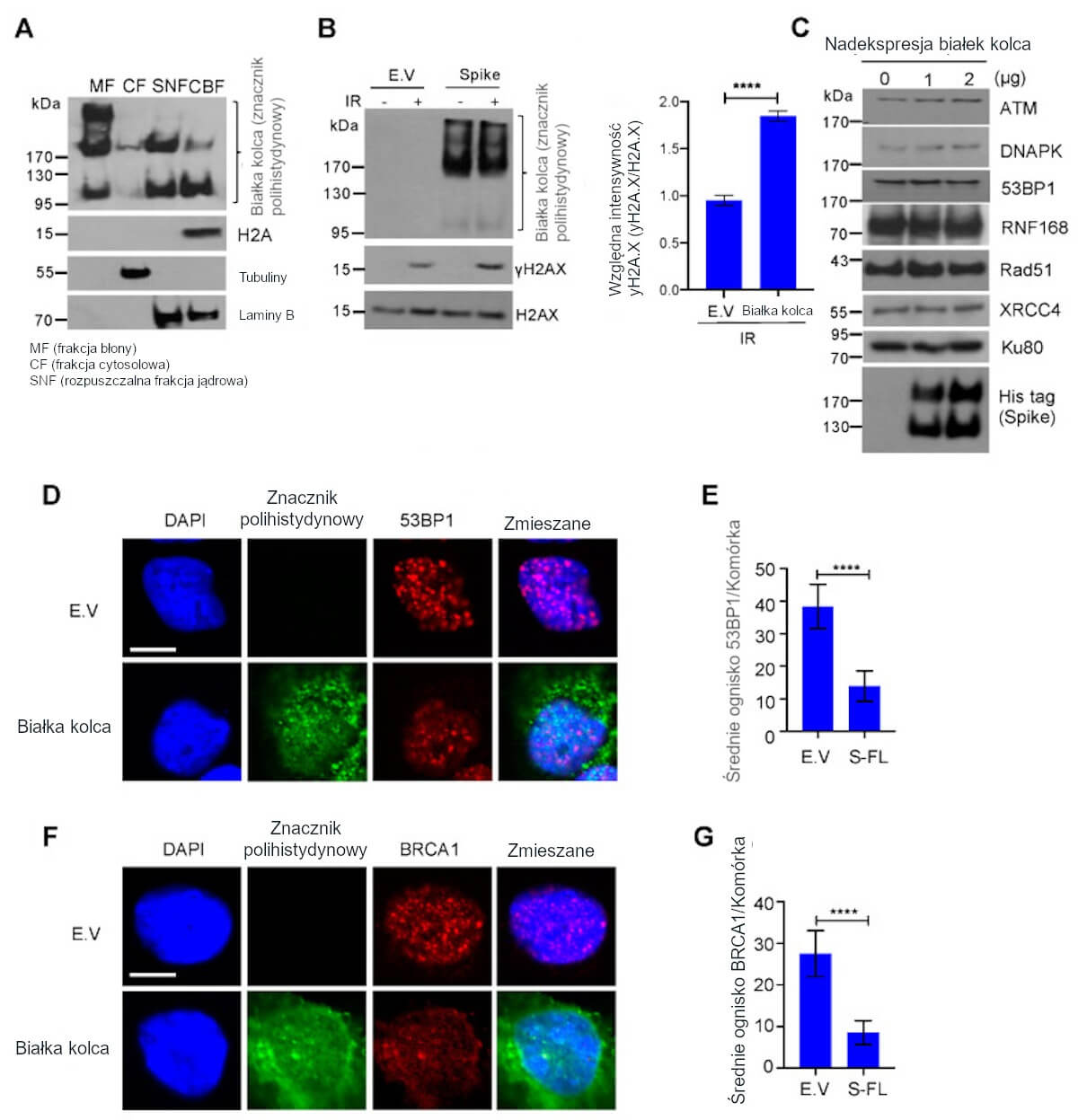
Białka kolca drugiego koronawirusa ciężkiego ostrego zespołu oddechowego (SARS-CoV-2) zakłócają rekrutację białek punktu kontrolnego pełniących funkcje naprawcze uszkodzeń DNA. (A) Frakcję błon (ang. membrane fraction, MF), frakcję cytosolową (ang. cytosolic fraction, CF), rozpuszczalną frakcję jądrową (ang. soluble nuclear fraction, SNF) i frakcję związaną z chromatyną (ang. chromatin-bound fraction, CBF) z komórek HEK293T transfekowanych białkami kolca wirusa SARS-CoV-2 poddano metodzie immunoblottingu w celu wykrycia białek kolca zawierających znacznik polihistydynowy oraz wskazane białka. (B) Ilustracja po lewej: Immunobloty markera uszkodzeń DNA γH2AX w komórkach HEK293T wykazujących ekspresję pustego wektora (E.V) i białek kolca po napromieniowaniu gamma o sile 10 Gy. Ilustracja po prawej: analogiczne ujęcie ilościowe immunoblotów po lewej. Wartości oznaczają średnie ± SD (n = 3). Istotność statystyczną określono stosując test T Studenta. **** p < 0.0001. (C) Immunobloty białek związanych z naprawą uszkodzeń DNA w komórkach HEK293T wykazujących ekspresję białek kolca. (D) Reprezentatywny obraz powstawania ognisk białek 53BP1 w komórkach HEK293 wykazujących ekspresję E.V i białek kolca narażonych na promieniowanie gamma o sile 10 Gy. Podziałka liniowa: 10 µm. (E) Analiza ilościowa ogniska 53BP1 na jądro. Wartości oznaczają średnią ± standardowy błąd średniej (ang. standard error of the mean, SEM), n = 50. (F) Powstawanie ognisk w komórkach HEK293 wykazujących ekspresję pustego wektora i białek kolca narażonych na promieniowanie gamma o sile 10 Gy. Podziałka liniowa: 10 µm. (G) Analiza ilościowa ogniska BRCA1 na jądro. Wartości oznaczają średnią ± SEM, n = 50. Istotność statystyczną określono stosując test T Studenta. **** p < 0.0001.
3.4. Białka kolca osłabiają rekombinację V(D)J
Proces naprawy uszkodzeń DNA, w szczególności ścieżka naprawy NHEJ, jest konieczny dla rekombinacji V(D)J, która leży u podstaw odporności limfocytów B i T[9]. Do dnia dzisiejszego wiele zatwierdzonych szczepionek przeciwko SARS-CoV-2, takich jak szczepionki mRNA i szczepionki adenowirusowe przeciwko COVID-19, opracowuje się w oparciu o białka kolca pełnej długości[25]. Chociaż to czy SARS-CoV-2 bezpośrednio zakaża prekursory limfocytów[26,27] pozostaje tematem dyskusji, niektóre świadectwa dowodzą, że zainfekowane komórki wydzielają egzosomy, które mogą dostarczać RNA lub białka SARS-CoV-2 do komórek docelowych[28,29]. Zbadaliśmy następnie czy białka kolca ograniczyły rekombinację V(D)J zależną od NHEJ. W tym celu opracowaliśmy system reporterowy in vitro rekombinacji V(D)J zgodnie z przeprowadzonym wcześniej badaniem[18] (Ilustracja S5). W porównaniu z pustym wektorem, nadekspresja białek kolca blokowała rekombinację V(D)J zależną od RAG w tym systemie reporterowym in vitro (Ilustracja 4).
Białka kolca osłabiają rekombinację V(D)J in vitro. (A) Schemat systemu reporterowego rekombinacji V(D)J. (B) Reprezentatywne wykresy cytometrii przepływowej pokazują, że białka kolca wirusa SARS-CoV-2 zakłócają rekombinację V(D)J in vitro. (C) Analiza ilościowa względnej rekombinacji V(D)J. Wartości reprezentują średnie ± SD, n = 3. Istotność statystyczną określono stosując test T Studenta. **** p < 0.0001.
4. Omówienie
Dokonane przez nas ustalenia dostarczają dowodów, że białka kolca przejmują kontrolę nad mechanizmem naprawy uszkodzeń DNA oraz mechanizmem odporności adaptacyjnej in vitro. Przedstawiamy potencjalny mechanizm, za pomocą którego białka kolca mogą osłabiać odporność adaptacyjną utrudniając naprawę uszkodzeń DNA. Mimo że nie opublikowano żadnych dowodów, iż wirus SARS-CoV-2 może zakażać tymocyty lub komórki limfoidalne szpiku kostnego, przeprowadzony przez nas test reporterowy V(D)J in vitro pokazuje, że białka kolca silnie zakłócały rekombinację V(D)J. Zgodne z uzyskanymi wynikami obserwacje kliniczne także wykazują, że ryzyko ciężkiego przebiegu lub zgonu na skutek COVID-19 wzrasta wraz z wiekiem, zwłaszcza u osób starszych, które znajdują się w grupie największego ryzyka[22]. Przyczyną tego stanu rzeczy może być zdolność białek kolca wirusa SARS-CoV-2 do osłabiania systemu naprawy uszkodzeń DNA u starszych osób, a w rezultacie do hamowania rekombinacji V(D)J i odporności adaptacyjnej. Nasze dane, przeciwnie, dostarczają cennych szczegółów dotyczących działu podjednostek białek kolca w procesie naprawy uszkodzeń DNA, które wskazują, że szczepionki oparte o białka kolca pełnej długości mogą powstrzymywać rekombinację V(D)J w limfocytach B, co również pokrywa się z wynikami przeprowadzonego niedawno badania dowodzącymi, że szczepionka opracowana w oparciu o białka kolca pełnej długości indukowała niższe miana przeciwciał w porównaniu do preparatu opartego o domenę wiążącą receptory[28]. Sugeruje to, że zastosowanie epitopów antygenów białek kolca w szczepionce przeciwko SARS-CoV-2 może być bezpieczniejsze i bardziej skuteczne niż białek pełnej długości. Reasumując, zidentyfikowaliśmy jeden z potencjalnie istotnych mechanizmów supresji przez wirus SARS-CoV-2 mechanizmu odporności adaptacyjnej gospodarza. Co więcej, nasze ustalenia sugerują ponadto występowanie potencjalnych skutków ubocznych szczepionki opartej o białka kolca pełnej długości. Dokonanie to poprawi nasze zrozumienie patogenezy COVID-19 i dostarczy nowych strategii opracowywania bardziej skutecznych i bezpieczniejszych szczepionek.